BẢN TIN KHOA HỌC
Phân lập CaPCR1, một gen OFP quy định tính trạng đỉnh quả ớt (Capsicum annuum L.)
Nguồn: Tingting Liu, Yiping Dong, Shenting Gao, Yingjia Zhou, Dan Liu, Jubin Wang, Zhenya Liu, Yingtian Deng & Feng Li. 2024. Identification of CaPCR1, an OFP gene likely involved in pointed versus concave fruit tip regulation in pepper (Capsicum annuum L.) using recombinant inbred lines
Theoretical and Applied Genetics; 14 June 2024; vol. 137, article 161
Theoretical and Applied Genetics; 14 June 2024; vol. 137, article 161

CaPCR1 (Capana12g002165) là một gen ứng cử viên điều tiết tính trạng đỉnh quả ớt lỏm hoặc nhọn (concave/pointed tip shape).
Hình lõm của đỉnh quả trên cây ớt là chỉ thị của tính trạng rất nhạy cảm với stress khô hạn và nhiệt độ lạnh, dẫn đến kết quả trái ớt có đỉnh nhọn (pointed tip fruit), ảnh hưởng đến giá trị thương phẩm. Tuy nhiên, rất ít nghiên cứu về tiến trình phát triển “fruit tip” như vậy và các gen có tính chất điều tiết trong cây ớt. Ở đây, người ta tiến hành nghiên cứu quá trình phát triển của bầu noãn trước khi thụ phấn (anthesis), đặc biệt là quan sát sự thay đổi kiểu dáng của “ovary tip” (đỉnh bầu noãn) một cách chi tiết. Kết quả cho thấy rằng “fruit tip shape” sau cùng phù hợp với đỉnh bầu noãn trước khi thụ phấn, một dạng hình đầu lõm dần dần phát triển. Quần thể con lai cận giao tái tổ hợp F4 (RILs) được tạo ra để thực hiện bản đồ di truyền các gen điều tiết tính trạng “fruit tip shape” thông qua tổ hợp lai giữa giống ớt LRS và giống ớt SBS là những dòng ớt cận giao. Gen CaPCR1 (Capana12g002165), một họ gen OFP (OVATE Family Protein), định vị trên vùng ứng cử viên ở nhiễm sắc thể 12. Ba chỉ thị SNPs được tìm thấy trong chuỗi trình tự mã hóa protein CaPCR1 giữa giống SBS và LRS, nhưng chỉ có một SNP marker điều khiển biến thể amino acid. Những biến thể về trình tự, bao gồm sự thay thế base, mất đoạn và chèn đoạn, đều được tìm thấy trong vùng promoter của gen này. Mức độ biểu hiện gen CaPCR1 lớn hơn một cách có ý nghĩa tại bầu noãn có đỉnh lõm hơn là bầu noãn có đỉnh nhọn. Kết quả phân tích qRT‒PCR cho thấy gen CaPCR1 chủ yếu được biểu hiện trong gynoecium, placenta và vỏ quả ớt xanh, phù hợp với chức năng của nó trong bầu noãn và sự phát triển trái ớt. Kết quả cho thấy CaPCR1 là một gen ứng cử viên quy định tính trạng “fruit tip shape” của cây ớt.
Xem https://link.springer.com/article/10.1007/s00122-024-04675-0
Hình lõm của đỉnh quả trên cây ớt là chỉ thị của tính trạng rất nhạy cảm với stress khô hạn và nhiệt độ lạnh, dẫn đến kết quả trái ớt có đỉnh nhọn (pointed tip fruit), ảnh hưởng đến giá trị thương phẩm. Tuy nhiên, rất ít nghiên cứu về tiến trình phát triển “fruit tip” như vậy và các gen có tính chất điều tiết trong cây ớt. Ở đây, người ta tiến hành nghiên cứu quá trình phát triển của bầu noãn trước khi thụ phấn (anthesis), đặc biệt là quan sát sự thay đổi kiểu dáng của “ovary tip” (đỉnh bầu noãn) một cách chi tiết. Kết quả cho thấy rằng “fruit tip shape” sau cùng phù hợp với đỉnh bầu noãn trước khi thụ phấn, một dạng hình đầu lõm dần dần phát triển. Quần thể con lai cận giao tái tổ hợp F4 (RILs) được tạo ra để thực hiện bản đồ di truyền các gen điều tiết tính trạng “fruit tip shape” thông qua tổ hợp lai giữa giống ớt LRS và giống ớt SBS là những dòng ớt cận giao. Gen CaPCR1 (Capana12g002165), một họ gen OFP (OVATE Family Protein), định vị trên vùng ứng cử viên ở nhiễm sắc thể 12. Ba chỉ thị SNPs được tìm thấy trong chuỗi trình tự mã hóa protein CaPCR1 giữa giống SBS và LRS, nhưng chỉ có một SNP marker điều khiển biến thể amino acid. Những biến thể về trình tự, bao gồm sự thay thế base, mất đoạn và chèn đoạn, đều được tìm thấy trong vùng promoter của gen này. Mức độ biểu hiện gen CaPCR1 lớn hơn một cách có ý nghĩa tại bầu noãn có đỉnh lõm hơn là bầu noãn có đỉnh nhọn. Kết quả phân tích qRT‒PCR cho thấy gen CaPCR1 chủ yếu được biểu hiện trong gynoecium, placenta và vỏ quả ớt xanh, phù hợp với chức năng của nó trong bầu noãn và sự phát triển trái ớt. Kết quả cho thấy CaPCR1 là một gen ứng cử viên quy định tính trạng “fruit tip shape” của cây ớt.
Xem https://link.springer.com/article/10.1007/s00122-024-04675-0
Áp dụng “multi-omics” để nghiên cứu di truyền cây bắp
Nguồn: Baber Ali, Bertrand Huguenin-Bizot, Maxime Laurent, François Chaumont, Laurie C. Maistriaux, Stéphane Nicolas, Hervé Duborjal, Claude Welcker, François Tardieu, Tristan Mary-Huard, Laurence Moreau, Alain Charcosset, Daniel Runcie & Renaud Rincent. 2024. High-dimensional multi-omics measured in controlled conditions are useful for maize platform and field trait predictions. Theoretical and Applied Genetics; July 2024; vol.137; article 175

Thông tin của “transcriptomics” và “proteomics” là nền tảng kiến thức có thể giúp chúng ta dự đoán được ảnh hưởng tính cộng (additive) và tính không cộng (non-additive) của tính trạng di truyền số lượng trên đồng ruộng.
Biến đổi khí hậu ảnh hưởng cây trồng thông qua stress khô hạn, nhiệt độ nóng, và những thay đổi bất thường của mùa vụ đe dọa đến sản lượng cây trồng trên toàn cầu. Khả năng của cơ sở dữ liệu “multi-omics”, ví dụ như “transcripts” và “proteins”, phản ánh sự đáp ứng của cây trồng đối với các yếu tố khí hậu có thể được ghi nhận trong các mô hình dự đoán nhằm tối đa hóa nội dung cải tiến cây trồng. Thực hiện việc định tính “multi-omics” theo đánh giá ngoài đồng là thách thức vì chi phí rất lớn. Tuy nhiên, khả năng làm được việc đó trên những dòng giống cây trồng tham chiếu (reference genotypes) ở điều kiện ngoại cảnh được kiểm soát là khả thi. Sử dụng “omics” trên nền tảng đo lường được, người ta đã trắc nghiệm nhiều phương pháp tiếp cận khác nhau trên cơ sở “multi-omics”, thông qua mô phỏng toán MegaLMM (high dimensional linear mixed model) nhằm dự đoán kiểu gen đối với tính trạng lý thuyết và tính trạng ở trên đồng ruộng với tập đoàn giống bắp gồm 244 dòng bắp lai (maize hybrids). Người ta giả định có hai kịch bản xảy ra: thứ nhất là tổ hợp ưu thế lai mới được dự đoán (CV-NH), và thứ hai là tổ hơi ưu thế lai quan sát được dự đoán (CV-POH). Trong cả hai kịch bản này, tất cả hybrids được định tính nhờ “omics” theo lý thuyết. Người ta thấy “omics” có thể dự đoán được cả hai ảnh hưởng tương tác gen cộng tính và gen không cộng tính đối với tất cả tính trạng, dẫn đến khả năng dự đoán chính xác hơn GBLUP. Nó nêu bật tính hiệu quả của chúng trong nắm bắt các quy trình điều tiết liên quan đến tăng trưởng Đối với những tính trạng trên đồng ruộng, người ta thấy thành phân gen cộng tính của “omics” chỉ cải thiện được một chút về khả năng dự đoán con lai ưu thế lai mới (new hybrids) (CV-NH, model MegaGAO) và đối với dự đoán con lai “partially observed hybrids” (CV-POH, model GAOxW-BLUP) khi so sánh với phân tích GBLUP. Kết luận: đo lường theo “omics” trên đồng ruộng sẽ rất đáng quan tâm trong dự đoán năng suất nếu chi phí của “omics” giảm xuống đáng kể.
Xem https://link.springer.com/article/10.1007/s00122-024-04679-w
Cơ sở dữ liệu metagenomics của “16S rRNA” đối với cộng đồng vi khuẩn nội sinh cây điều (Anacardium occidentale L.) ở Dak Lak, Vietnam
Nguồn: Dinh Minh Tran, Thi Huyen Nguyen. 2024. 16S rRNA metagenomic dataset on endophytic bacterial community of the cashew plant (Anacardium occidentale L.) grown in Dak Lak Province of Vietnam. Data Brief ; 2024 Jan 9: 52:110039. doi: 10.1016/j.dib.2024.110039.

Hiện nay, Việt Nam là một trong những nước sản xuất điều và xuất khẩu điếu lớn nhất thế giới. Cây điều (Anacardium occidentale L.) là một trong những cây công nghiệp chủ lực của tỉnh Dak Lak, Vietnam. Hiểu được vi khuẩn nội sinh (endophytic bacteria) của cây điều, người ta có thể thành công trong sản xuất ra phân sinh học mới nhằm ổn định sản lượng hạt điều. Trong báo cáo này, người ta thu thập mẫu rễ điều trên đồng ruộng trong năm 2021 tại Dak Lak.
Sử dụng DNeasy Powersoil kit để tách chiết DNA của vi khuẩn nội sinh từ mẫu rễ điều. Gen 16S rRNA (V1-V9 regions) được khuếch đại bằng PCR, và thư viện của amplicons được chuẩn bị nhờ Swift amplicon 16S plus ITS panel kit. Áp dụng Illumina MiSeq platform để giải trình tự thự viện amplicon này thông qua 16S rRNA metagenomics. Phân loại cho thấy Gammaproteobacteria chiếm 38.77 % và Alphaproteobacteria chiếm 37.76 %; đó là những class chiếm ưu thế trong quần thể vi khuẩn nội sinh. Kết quả phân tích chức năng cho thấy chức năng sinh tổng hợp (72.78 %) là chức năng chủ lực của cộng đồng vi khuẩn nội sinh này. Trình tự bản thảo (Fastq files) được gửi vào Mendeley Data. Cơ sở dữ liệu này cung cấp cách nhìn nhận có chiều sâu về cộng đồng vi khuẩn nội sinh này trong cây điều ở tỉnh Dak Lak. Số liệu rất có giá trị nhằm phát triển sau này loại hình phân bón sinh học trong canh tác điều cho năng suất ổn định. Đây là báo cáo đầu tiên về cộng đồng vi khuẩn nội sinh trên cây điều được trồng tại tỉnh Dak Lak, Tây Nguyên Việt Nam.
Xem https://pubmed.ncbi.nlm.nih.gov/38293582/
Sử dụng DNeasy Powersoil kit để tách chiết DNA của vi khuẩn nội sinh từ mẫu rễ điều. Gen 16S rRNA (V1-V9 regions) được khuếch đại bằng PCR, và thư viện của amplicons được chuẩn bị nhờ Swift amplicon 16S plus ITS panel kit. Áp dụng Illumina MiSeq platform để giải trình tự thự viện amplicon này thông qua 16S rRNA metagenomics. Phân loại cho thấy Gammaproteobacteria chiếm 38.77 % và Alphaproteobacteria chiếm 37.76 %; đó là những class chiếm ưu thế trong quần thể vi khuẩn nội sinh. Kết quả phân tích chức năng cho thấy chức năng sinh tổng hợp (72.78 %) là chức năng chủ lực của cộng đồng vi khuẩn nội sinh này. Trình tự bản thảo (Fastq files) được gửi vào Mendeley Data. Cơ sở dữ liệu này cung cấp cách nhìn nhận có chiều sâu về cộng đồng vi khuẩn nội sinh này trong cây điều ở tỉnh Dak Lak. Số liệu rất có giá trị nhằm phát triển sau này loại hình phân bón sinh học trong canh tác điều cho năng suất ổn định. Đây là báo cáo đầu tiên về cộng đồng vi khuẩn nội sinh trên cây điều được trồng tại tỉnh Dak Lak, Tây Nguyên Việt Nam.
Xem https://pubmed.ncbi.nlm.nih.gov/38293582/
Đa dạng di truyền và kiến trúc quần thể các mẫu giống hồ tiêu Piper nigrum trên cơ sở chỉ thị phân tử SNP và giải trình tự thế hệ mới (NGS)

Nguồn: Nilni A Wimalarathna, Anushka M Wickramasuriya, Dominik Metschina, Luiz A Cauz-Santos, Dharshani Bandupriya, Kahandawa G S U Ariyawansa, Bhathiya Gopallawa, Mark W Chase, Rosabelle Samuel , Tara D Silva. 2024. Genetic diversity and population structure of Piper nigrum (black pepper) accessions based on next-generation SNP markers. PlosOne 2024 Jun 26;19(6):e0305990. doi: 10.1371/journal.pone.0305990.
Mặc dù gía trị kinh tế của Piper nigrum (black pepper) rất cao, trên thương trường quốc tế, nhưng việc phát triển và sử dụng tài nguyên di truyền của hệ gen này (genomic resources) vẫn còn nhiều hạn chế, với đánh giá đa dạng di truyền thường thường chỉ dựa vào một vài mẫu hoặc chỉ thị phân tử DNA Ở đây, người ta tiến hành tiếp cận phương pháp giải trình tự DNA gắn với vị trí hạn chế (restriction-site associated DNA sequencing) nhằm phân tích 175 mẫu giống cây P. nigrum thu thập ở 8 vùng trồng hồ tiêu chủ lực tại Sri Lanka. Kết quả chạy trình tự DNA cho thấy có 1.976 triệu “reads” ban đầu, trung bình đạt 11,3 triệu “reads” trên mỗi mẫu giống tiêu đen, biểu trưng cho 150.356 chỉ thị SNPs chất lượng đa hình tốt, phân bố trên 26 nhiễm sắc thể. Phân tích kiến trúc quần thể cho thấy có hai “subpopulations” (K = 2): một nhóm biểu thị tính “dominant” với 152 mẫu giống tiêu thu thập từ vườn nhà (home gardens) và từ đồng ruộng đại trà (large-scale cultivations), một nhóm khác nhỏ hơn có 23 mẫu giống tiêu từ bộ sưu tập bản địa của vườn nhà (home gardens). Sự phân nhóm như vậy (clustering) được hỗ trợ của phân tích PCA, với 2 components đầu tiên giải thích được 35,2 và 12,1% biến thiên kiểu hình. Phân tích đa dạng di truyền cho thấy có biểu hiện của “substantial gene flow” (Nm = 342,21) và chỉ số ổn định FST thấp (low fixation index) = 0.00073, giữa hai subpopulations này, sự phân hóa các chức năng di truyền không rõ ràng trong các mẫu giống tiêu có nguồn gốc sinh thái canh tác khác nhau. Kết quả chứng minh rằng hầu hết các giống tiêu hiện nay được trồng ở Sri Lanka đều chia sẻ từ một nền tảng di truyền chung, cho thấy sự cần thiết phải mở rộng hơn nữa nền tảng di truyền, mới có thể tăng cường sức chịu đựng của chúng với stress sinh học và phi sinh học. Đây là nghiên cứu đầu tiên phân tích đa dạng di truyền cây hồ tiêu thông qua chỉ thị SNP phân giải cao, đặt nền tảng cho nghiên cứu tổng hợp trong tương lai về “genome-wide association” trên cơ sở phát hiện gen nhờ chỉ thị SNP và cải tiến giống hồ tiêu sắp tới.
Xem https://pubmed.ncbi.nlm.nih.gov/38924027/
Xem https://pubmed.ncbi.nlm.nih.gov/38924027/
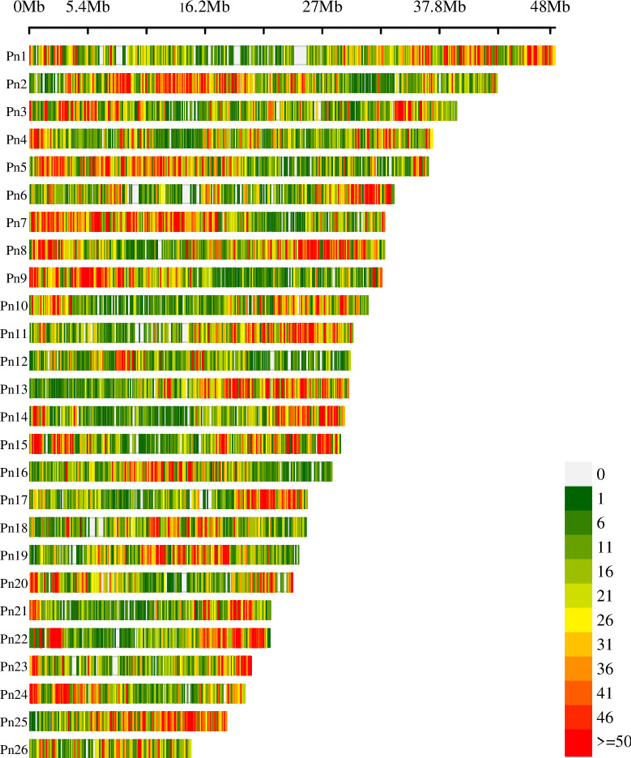
Hình: Mật độ chỉ thị phân tử SNP. Sự phân bố của 150.329 SNP markers trong cửa sổ có kích thước 0,1 Mb định vị trên 26 nhiễm sắc thể. Thanh có màu sắc biểu thị số SNP được đếm trong quãng 0,1 Mb.
.png)






.png)









